To build up a library is to create a life. It’s never just a random collection of books.
Z-Library
1
Suivez ce lien ou recherchez le bot "@BotFather" sur Telegram
2
Envoyer la commande /newbot

3
Entrez un nom pour votre bot

4
Spécifiez le nom d'utilisateur pour le bot
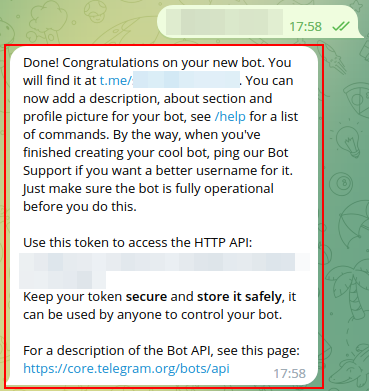
5
Copier le dernier message de BotFather et le coller ici